6.10 EMP_WGCNA_cor_analysis
在WGCNA聚类分析后,模块EMP_WGCNA_cor_analysis使用PCA方法对聚类模块进行降维,将解释度最大的轴与目标数据进行关联分析,从而探究哪些聚类数据具有潜在的后续分析价值。
6.10.1 基于单一组学与样本相关数据的WGCNA分析
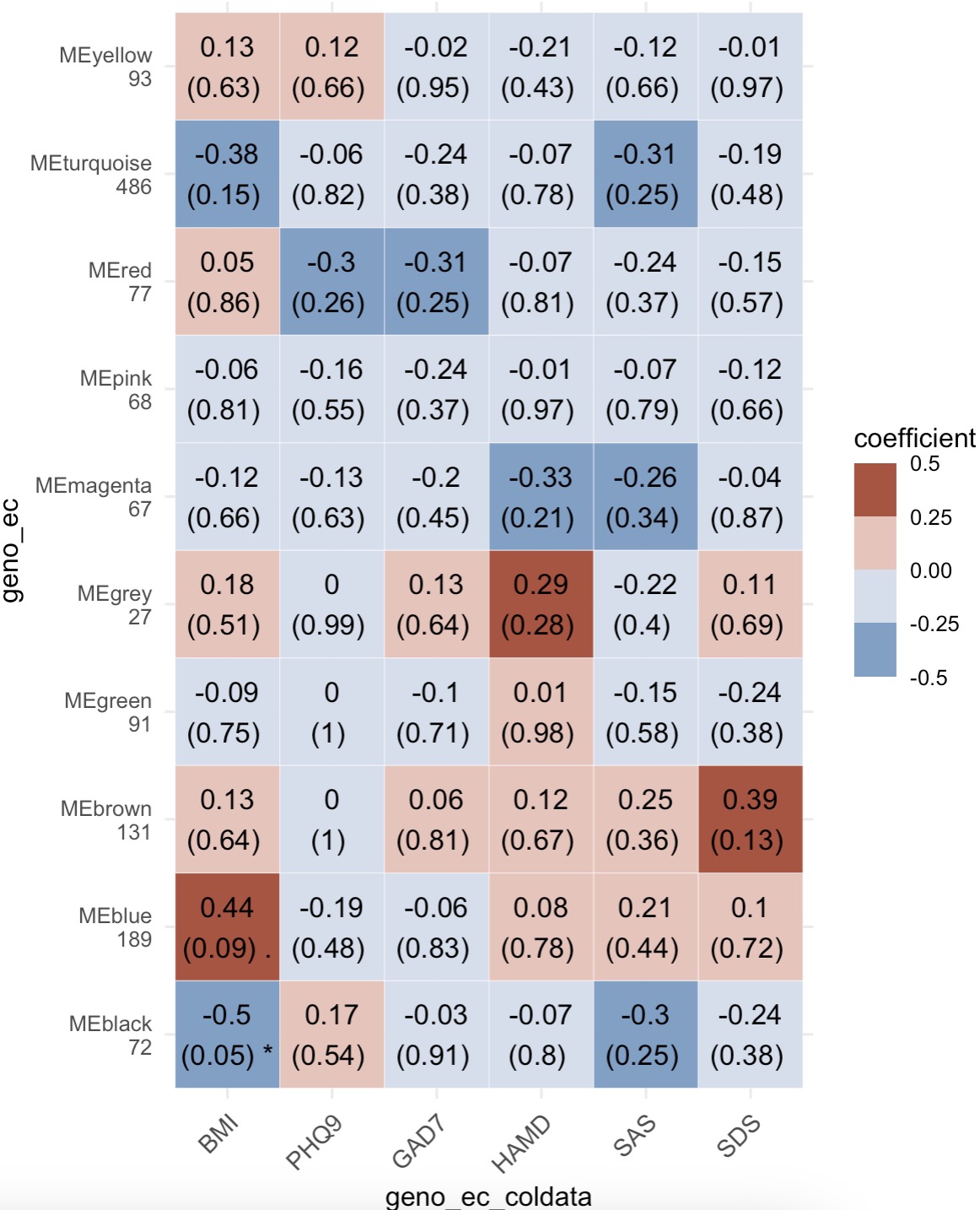
🏷️示例1:
从MAE对象中提取组学项目geno_ec的assay,使用edgeR的方法筛选核心微生物,利用WGCNA进行聚类分析并与样本相关数据进行关联分析,通过绘制热图进行结果可视化。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_identify_assay(method = 'edgeR',estimate_group = 'Group') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85,mergeCutHeight=0.4) |>
EMP_WGCNA_cor_analysis(coldata_to_assay = c('BMI','PHQ9','GAD7','HAMD','SAS','SDS')) |>
EMP_heatmap_plot()
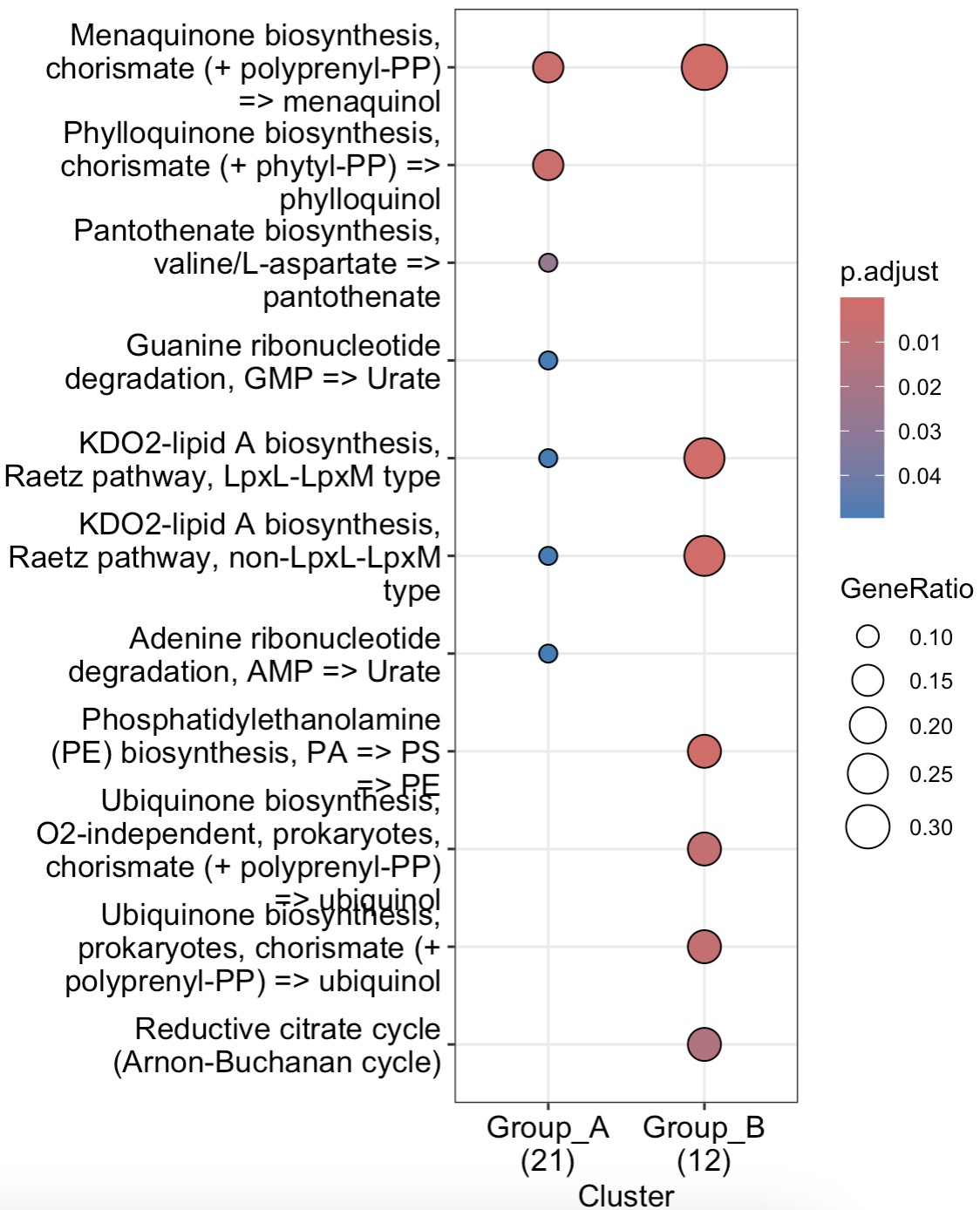
🏷️示例2:
在利用WGCNA进行聚类分析并与样本相关数据进行关联分析后,可选择与目标样本相关数据相关性具有统计学意义的模块进行下游的差异性分析和富集分析。
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_identify_assay(method = 'edgeR',estimate_group = 'Group') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85,mergeCutHeight=0.4) |>
EMP_WGCNA_cor_analysis(coldata_to_assay = c('BMI','PHQ9','GAD7','HAMD','SAS','SDS')) |>
EMP_filter(feature_condition = WGCNA_color=='black') |>
EMP_diff_analysis(method = 'DESeq2',.formula = ~Group) |>
EMP_enrich_analysis(keyType = 'ec',KEGG_Type = 'MKEGG',pvalueCutoff=0.05) |>
EMP_enrich_dotplot()
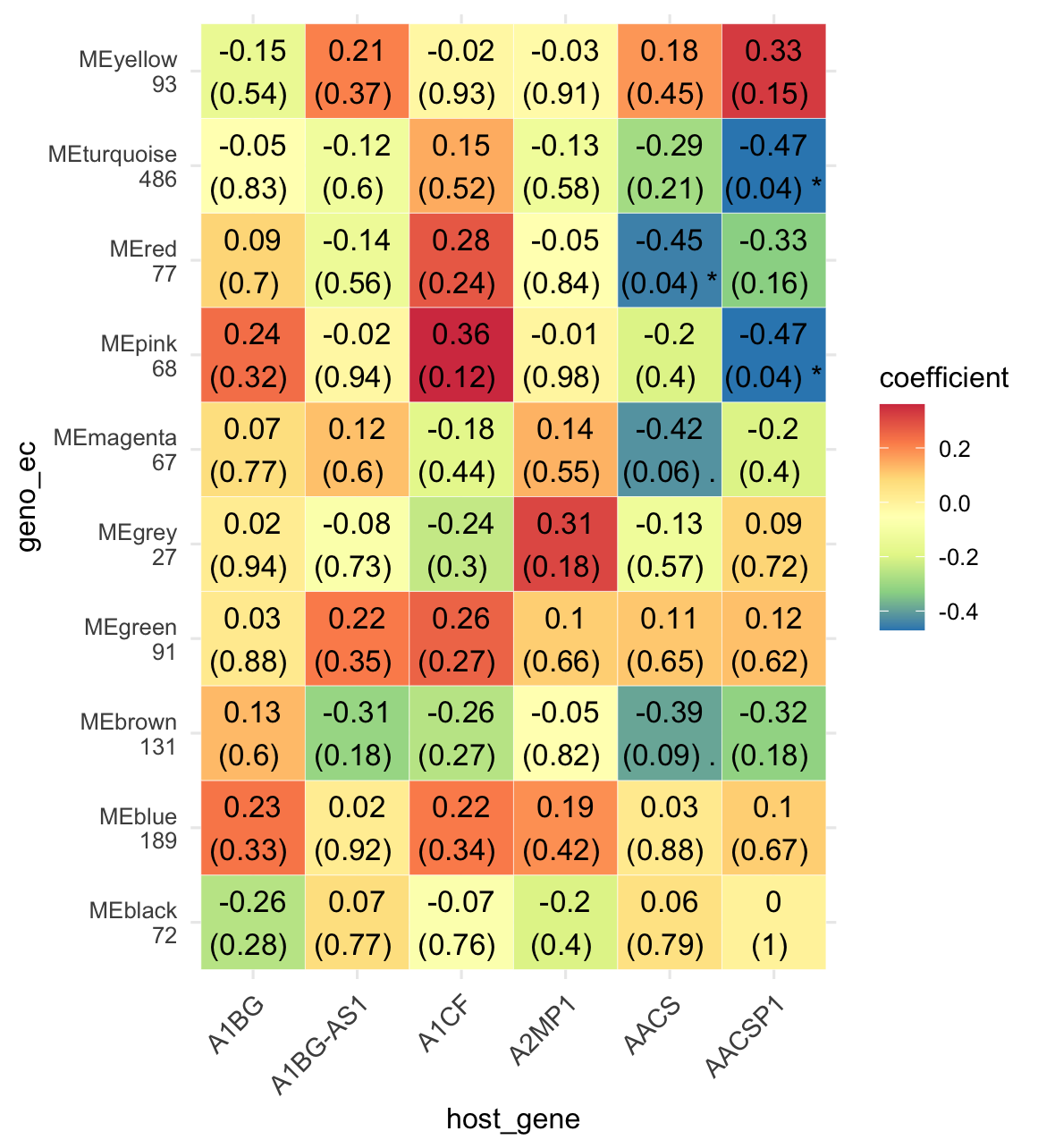
6.10.2 基于多个组学数据的WGCNA分析
🏷️示例:
首先,从MAE对象中提取组学项目geno_ec的assay,使用edgeR的方法筛选差异基因,并进行WGCNA聚类分析。其次,从MAE对象中提取组学项目host_gene的assay中的感兴趣基因(或使用差异性分析等方法选择目标基因)。 使用+符号连接两个组学,并进行WGCNA相关性分析。
注意:
在此模式下,默认会从第一个组学中提取WGCNA聚类结果与第二个组学进行WGCNA相关分析,因此注意使用
在此模式下,默认会从第一个组学中提取WGCNA聚类结果与第二个组学进行WGCNA相关分析,因此注意使用
+合并组学的顺序。
k1 <- MAE |>
EMP_assay_extract('geno_ec') |>
EMP_identify_assay(method = 'edgeR',estimate_group = 'Group') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85,mergeCutHeight=0.4)
k2 <- MAE |>
EMP_assay_extract('host_gene',pattern = c('A1BG','A1CF','A2MP1','AACS'),
pattern_ref = 'feature')
(k1 + k2) |>
EMP_WGCNA_cor_analysis() |>
EMP_heatmap_plot(palette = 'Spectral')